Conformers:
ABBIImiBZICOPNSYNN
Click the Summary/Torsions/Similar/... tabs for more details.
AMP RNA APTAMER COMPLEX, NMR, 8 STRUCTURES
Click a row in table or a step in viewer for analysis of results. Click column headers to sort data.
Steps with non-standard or missing atoms have not been assigned, description of conformers is defined in the table.
Results of the assignment of 112 detected steps in 8 model(s), can be also downloaded as csv or json file. Average confal 0, percentile 0.
Click a row in table or a step in viewer for analysis of results. Click column headers to sort data.
Define restraints for steps within Å cartesian RMSD, global sigma scaling factor or use per step sigma scaling.
| step number | Step name | CANA | NtC | confal | rmsd |
|---|---|---|---|---|---|
| 001 | 1am0-m1_A_G6_G7 | NAN | NANT | 0 | 1.12 |
| 002 | 1am0-m2_A_G6_G7 | NAN | NANT | 0 | 1.11 |
| 003 | 1am0-m3_A_G6_G7 | NAN | NANT | 0 | 1.11 |
| 004 | 1am0-m4_A_G6_G7 | NAN | NANT | 0 | 1.09 |
| 005 | 1am0-m5_A_G6_G7 | NAN | NANT | 0 | 1.29 |
| 006 | 1am0-m6_A_G6_G7 | NAN | NANT | 0 | 1.66 |
| 007 | 1am0-m7_A_G6_G7 | NAN | NANT | 0 | 0.65 |
| 008 | 1am0-m8_A_G6_G7 | NAN | NANT | 0 | 0.51 |
| 009 | 1am0-m1_A_G7_G8 | NAN | NANT | 0 | 0.81 |
| 010 | 1am0-m2_A_G7_G8 | NAN | NANT | 0 | 0.93 |
| 011 | 1am0-m3_A_G7_G8 | NAN | NANT | 0 | 0.96 |
| 012 | 1am0-m4_A_G7_G8 | NAN | NANT | 0 | 1.15 |
| 013 | 1am0-m5_A_G7_G8 | NAN | NANT | 0 | 0.94 |
| 014 | 1am0-m6_A_G7_G8 | NAN | NANT | 0 | 0.86 |
| 015 | 1am0-m7_A_G7_G8 | NAN | NANT | 0 | 1.10 |
| 016 | 1am0-m8_A_G7_G8 | NAN | NANT | 0 | 0.89 |
| 017 | 1am0-m1_A_G8_A9 | NAN | NANT | 0 | 1.05 |
| 018 | 1am0-m2_A_G8_A9 | NAN | NANT | 0 | 1.07 |
| 019 | 1am0-m3_A_G8_A9 | NAN | NANT | 0 | 1.07 |
| 020 | 1am0-m4_A_G8_A9 | NAN | NANT | 0 | 0.53 |
| 021 | 1am0-m5_A_G8_A9 | NAN | NANT | 0 | 0.46 |
| 022 | 1am0-m6_A_G8_A9 | NAN | NANT | 0 | 0.81 |
| 023 | 1am0-m7_A_G8_A9 | NAN | NANT | 0 | 1.94 |
| 024 | 1am0-m8_A_G8_A9 | NAN | NANT | 0 | 1.25 |
| 025 | 1am0-m1_A_A9_A10 | NAN | NANT | 0 | 0.67 |
| 026 | 1am0-m2_A_A9_A10 | NAN | NANT | 0 | 0.69 |
| 027 | 1am0-m3_A_A9_A10 | NAN | NANT | 0 | 0.68 |
| 028 | 1am0-m4_A_A9_A10 | AAA | AA08 | 42 | 0.45 |
| 029 | 1am0-m5_A_A9_A10 | NAN | NANT | 0 | 0.83 |
| 030 | 1am0-m6_A_A9_A10 | NAN | NANT | 0 | 0.62 |
| 031 | 1am0-m7_A_A9_A10 | NAN | NANT | 0 | 1.26 |
| 032 | 1am0-m8_A_A9_A10 | NAN | NANT | 0 | 0.95 |
| 033 | 1am0-m1_A_A10_G11 | NAN | NANT | 0 | 0.82 |
| 034 | 1am0-m2_A_A10_G11 | NAN | NANT | 0 | 0.82 |
| 035 | 1am0-m3_A_A10_G11 | NAN | NANT | 0 | 0.77 |
| 036 | 1am0-m4_A_A10_G11 | NAN | NANT | 0 | 0.81 |
| 037 | 1am0-m5_A_A10_G11 | NAN | NANT | 0 | 2.35 |
| 038 | 1am0-m6_A_A10_G11 | NAN | NANT | 0 | 0.82 |
| 039 | 1am0-m7_A_A10_G11 | NAN | NANT | 0 | 0.92 |
| 040 | 1am0-m8_A_A10_G11 | NAN | NANT | 0 | 0.98 |
| 041 | 1am0-m1_A_G11_A12 | NAN | NANT | 0 | 1.32 |
| 042 | 1am0-m2_A_G11_A12 | NAN | NANT | 0 | 1.31 |
| 043 | 1am0-m3_A_G11_A12 | NAN | NANT | 0 | 1.29 |
| 044 | 1am0-m4_A_G11_A12 | NAN | NANT | 0 | 1.34 |
| 045 | 1am0-m5_A_G11_A12 | NAN | NANT | 0 | 1.35 |
| 046 | 1am0-m6_A_G11_A12 | NAN | NANT | 0 | 1.35 |
| 047 | 1am0-m7_A_G11_A12 | NAN | NANT | 0 | 1.30 |
| 048 | 1am0-m8_A_G11_A12 | NAN | NANT | 0 | 1.29 |
| 049 | 1am0-m1_A_A12_A13 | NAN | NANT | 0 | 1.63 |
| 050 | 1am0-m2_A_A12_A13 | NAN | NANT | 0 | 1.78 |
| 051 | 1am0-m3_A_A12_A13 | NAN | NANT | 0 | 1.81 |
| 052 | 1am0-m4_A_A12_A13 | NAN | NANT | 0 | 1.86 |
| 053 | 1am0-m5_A_A12_A13 | NAN | NANT | 0 | 1.50 |
| 054 | 1am0-m6_A_A12_A13 | NAN | NANT | 0 | 1.47 |
| 055 | 1am0-m7_A_A12_A13 | NAN | NANT | 0 | 1.78 |
| 056 | 1am0-m8_A_A12_A13 | NAN | NANT | 0 | 1.44 |
| 057 | 1am0-m1_A_A13_A14 | NAN | NANT | 0 | 0.68 |
| 058 | 1am0-m2_A_A13_A14 | NAN | NANT | 0 | 0.58 |
| 059 | 1am0-m3_A_A13_A14 | NAN | NANT | 0 | 0.66 |
| 060 | 1am0-m4_A_A13_A14 | NAN | NANT | 0 | 0.94 |
| 061 | 1am0-m5_A_A13_A14 | NAN | NANT | 0 | 0.73 |
| 062 | 1am0-m6_A_A13_A14 | NAN | NANT | 0 | 0.69 |
| 063 | 1am0-m7_A_A13_A14 | NAN | NANT | 0 | 1.23 |
| 064 | 1am0-m8_A_A13_A14 | NAN | NANT | 0 | 0.90 |
| 065 | 1am0-m1_A_A14_C15 | NAN | NANT | 0 | 0.58 |
| 066 | 1am0-m2_A_A14_C15 | NAN | NANT | 0 | 0.64 |
| 067 | 1am0-m3_A_A14_C15 | AAA | AA00 | 11 | 0.50 |
| 068 | 1am0-m4_A_A14_C15 | A-B | AB05 | 9 | 0.55 |
| 069 | 1am0-m5_A_A14_C15 | AAA | AA00 | 38 | 0.40 |
| 070 | 1am0-m6_A_A14_C15 | NAN | NANT | 0 | 0.69 |
| 071 | 1am0-m7_A_A14_C15 | NAN | NANT | 0 | 1.15 |
| 072 | 1am0-m8_A_A14_C15 | NAN | NANT | 0 | 1.60 |
| 073 | 1am0-m1_A_C15_U16 | NAN | NANT | 0 | 0.98 |
| 074 | 1am0-m2_A_C15_U16 | NAN | NANT | 0 | 0.85 |
| 075 | 1am0-m3_A_C15_U16 | NAN | NANT | 0 | 0.67 |
| 076 | 1am0-m4_A_C15_U16 | NAN | NANT | 0 | 0.93 |
| 077 | 1am0-m5_A_C15_U16 | NAN | NANT | 0 | 0.89 |
| 078 | 1am0-m6_A_C15_U16 | NAN | NANT | 0 | 0.98 |
| 079 | 1am0-m7_A_C15_U16 | NAN | NANT | 0 | 0.55 |
| 080 | 1am0-m8_A_C15_U16 | NAN | NANT | 0 | 0.60 |
| 081 | 1am0-m1_A_U16_G17 | AAA | AA08 | 33 | 0.48 |
| 082 | 1am0-m2_A_U16_G17 | NAN | NANT | 0 | 0.70 |
| 083 | 1am0-m3_A_U16_G17 | NAN | NANT | 0 | 0.66 |
| 084 | 1am0-m4_A_U16_G17 | NAN | NANT | 0 | 0.60 |
| 085 | 1am0-m5_A_U16_G17 | NAN | NANT | 0 | 0.57 |
| 086 | 1am0-m6_A_U16_G17 | NAN | NANT | 0 | 1.39 |
| 087 | 1am0-m7_A_U16_G17 | NAN | NANT | 0 | 1.32 |
| 088 | 1am0-m8_A_U16_G17 | AAA | AA08 | 62 | 0.50 |
| 089 | 1am0-m1_A_G17_U18 | NAN | NANT | 0 | 0.56 |
| 090 | 1am0-m2_A_G17_U18 | AAA | AA00 | 19 | 0.41 |
| 091 | 1am0-m3_A_G17_U18 | NAN | NANT | 0 | 1.27 |
| 092 | 1am0-m4_A_G17_U18 | NAN | NANT | 0 | 1.30 |
| 093 | 1am0-m5_A_G17_U18 | NAN | NANT | 0 | 1.38 |
| 094 | 1am0-m6_A_G17_U18 | NAN | NANT | 0 | 1.44 |
| 095 | 1am0-m7_A_G17_U18 | NAN | NANT | 0 | 1.12 |
| 096 | 1am0-m8_A_G17_U18 | NAN | NANT | 0 | 1.24 |
| 097 | 1am0-m1_A_A33_G34 | NAN | NANT | 0 | 0.59 |
| 098 | 1am0-m2_A_A33_G34 | NAN | NANT | 0 | 1.10 |
| 099 | 1am0-m3_A_A33_G34 | NAN | NANT | 0 | 1.50 |
| 100 | 1am0-m4_A_A33_G34 | NAN | NANT | 0 | 1.01 |
| 101 | 1am0-m5_A_A33_G34 | NAN | NANT | 0 | 0.82 |
| 102 | 1am0-m6_A_A33_G34 | NAN | NANT | 0 | 0.84 |
| 103 | 1am0-m7_A_A33_G34 | NAN | NANT | 0 | 1.40 |
| 104 | 1am0-m8_A_A33_G34 | NAN | NANT | 0 | 1.41 |
| 105 | 1am0-m1_A_G34_C35 | NAN | NANT | 0 | 1.60 |
| 106 | 1am0-m2_A_G34_C35 | NAN | NANT | 0 | 1.30 |
| 107 | 1am0-m3_A_G34_C35 | NAN | NANT | 0 | 0.84 |
| 108 | 1am0-m4_A_G34_C35 | NAN | NANT | 0 | 1.10 |
| 109 | 1am0-m5_A_G34_C35 | NAN | NANT | 0 | 1.01 |
| 110 | 1am0-m6_A_G34_C35 | NAN | NANT | 0 | 1.90 |
| 111 | 1am0-m7_A_G34_C35 | NAN | NANT | 0 | 1.61 |
| 112 | 1am0-m8_A_G34_C35 | NAN | NANT | 0 | 1.56 |
Steps with non-standard or missing atoms have not been assigned, description of conformers is defined in the table.
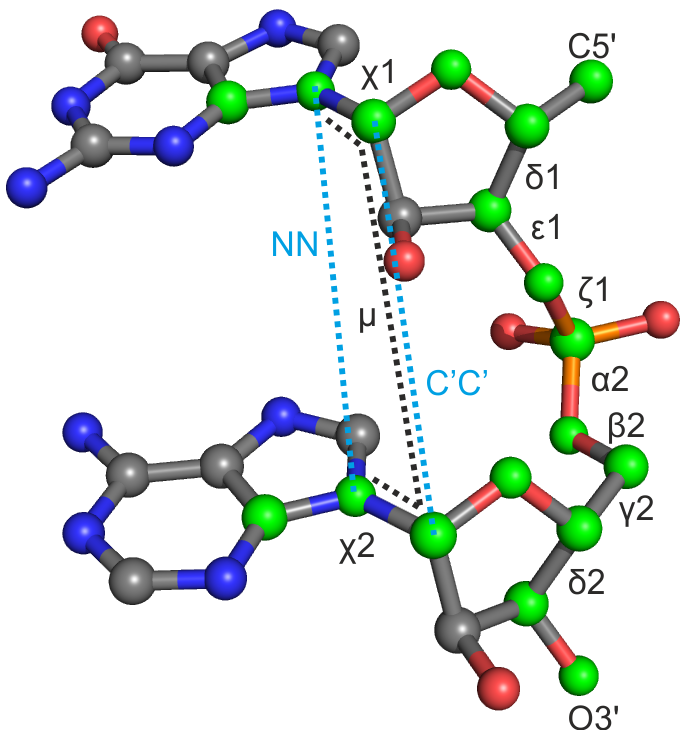
|
step: Table of conformers |
| δ1 | ε1 | ζ1 | α2 | β2 | γ2 | δ2 | χ1 | χ2 | μ | NN | CC | |
| step_torsions | ||||||||||||
| ntC_average | ||||||||||||
| Δ torsions | ||||||||||||
| torsion scores |
| comments | step confal = NaN cartesian RMSD = NaN Å pseudorotation: NA details: NA |
Similarity of step to class averages
Download
Results as csv or json file.Best NtC fitted to input structure.
Restraints
Restraints file for REFMAC (steps with RMSD <= 0.5Å)Commands file for MMB (steps with RMSD <= 0.5Å)
Restraints file for Phenix (steps with RMSD <= 0.5Å) [?]
Definitions
Average parameters (csv), esd values (csv), and Cartesian coordinates of conformers.Download the papers
Description of DNATCO server:Černý et al., Nucleic Acids Research, 44, W284 (2016).
Definition of conformers:
Schneider et al., Acta Cryst D, 74, 52-64 (2018).
Example of application:
Schneider et al., Genes, 8(10), 278, (2017).
Browse conformers
Show
| Color by conformation group (pyramids) | |
|---|---|
| group | |||||||||
| visible |
| Color by NtC (balls) |
|---|
| A | ||||
|---|---|---|---|---|
| A-B | ||||
|---|---|---|---|---|
| B-A | ||||
|---|---|---|---|---|
| B | ||||
|---|---|---|---|---|
| IC | ||||
|---|---|---|---|---|
| OPN | ||||
|---|---|---|---|---|
| Z | ||||
|---|---|---|---|---|
| N | ||||
|---|---|---|---|---|